注意:
- 本指南展示的只是 simmr 的一个非常基本的应用,您可以将其作为一个入门指南。如需深入了解这一程序,请参阅 simmr 官网提供的操作指南。
- 本文只介绍 simmr 的使用方法,不深入讨论其原理(因为我也看不明白……)。
- 本文是笔者摸鱼的时候写的,里面会有很多不完善的地方,欢迎大家批评指正。日后笔者可能还会对本文进行更新,但考虑到近期比较紧张的工作安排,大概率是短期内不会更新了。
- 本文的内容……说实话是比较专业的,对路人读者可能不太友好。只能这么说,需要用的人自然会懂,看不懂的人大概率也不需要懂

目的与情景导入
根据样品的稳定同位素组成信息,分析样品中有机物的来源。比如:
- 根据河水中硝酸盐的氮、氧同位素信息,确认该河流中硝酸盐的来源(土壤中的含氮有机物?生活污水?化肥?)
- 根据某海带养殖区海底的淤泥的碳、氮同位素信息,确认淤泥中有机物的来源(海洋中的浮游植物?陆生C3植物?陆生C4植物?海带产生的碎屑?)
数据测量
在开始分析前,我们需要测定样品的稳定同位素组成,比如碳稳定同位素 δ13C、氮稳定同位素组成 δ15N、氧稳定同位素 δ18O、氢同位素 δD。
各参数的测量方法不尽相同,这里不展开介绍。
环境准备
1 安装 R 语言
分析有机物来源的程序 simmr 是一个 R 语言程序包。使用它需要您先安装 R 语言。不同操作系统安装 R 语言的方法请参考:
2 安装 JAGS
simmr 工作时需要调用 JAGS 这个计算工具。在 Ubuntu、Debian 系统中,可执行如下命令安装 JAGS:
sudo apt install r-cran-rjags
对于 Windows 系统,请到这里下载 JAGS 的安装包(下载最新版的 JAGS-xxx.exe 即可,xxx 表示安装程序的版本号):
3 安装 simmr 和 showfont
进入 R shell,执行如下命令:
install.packagse('simmr')
install.packagse('showfont')
其中,
simmr是分析物质来源的程序,也是本指南的核心showfont用于在作图时正确显示汉字
准备工作空间
接下来的演示中,我们以使用 δ13C 和 δ15N 两种稳定同位素为例开展物质来源分析进行演示。
我们需要新建一个文件夹,作为工作空间。该文件夹中需要包含如下内容:
mixture.csv:存放样品的稳定同位素组成信息。该文件包含 3 列,分别为- group:样品的分组依据,为正整数
- d13C:样品的 δ13C 值
- d15N:样品的 δ15N 值
- 后面几列可以根据您实际使用的稳定同位素进行替换,如 d18O、dH 等。
- 除了稳定同位素比值,也可以使用元素含量比值,如碳氮摩尔比(C/N),但在命名的时候需要避免使用特殊符号,因此您可以将其写成 CvsN
sources.csv:存放各物质来源(下文称“端元”)的稳定同位素组成信息。该文件包含 5 列,分别为- Sources:端元名称
- Meand13C:某一端元的 δ13C 值
- SDd13C:某一端元的 δ13C 值的不确定度
- Meand15N:某一端元的 δ15N 值
- SDd15N:某一端元的 δ15N 值的不确定度
- 这里的 d13C、d15N 同样可以根据研究需要做适当调整,注意要和
mixture.csv相对应。
myfont.ttf:一个支持汉字的字体文件,要求是 ttf 格式simmr.R:R 语言脚本,用于执行分析程序
编写脚本
编辑工作文件夹下的 simmr.R,输入如下代码。每一行代码是干什么的,我都用尽可能详细的注释进行了说明。大家可以根据自己的需要酌情调整。
#!/usr/bin/Rscript
library(simmr)
library(showtext) # 该包用于在图表中显示汉字
showtext_auto() # 让程序自动处理汉字等其他字符,保障所有文本正常显示
font_add("myfont", "myfont.ttf") # 在工作目录下放一个支持中文的字体文件 myfont.ttf,在绘图时使用
# 1 加载数据
# 这里用的是软件包中提供的示例数据
mydata <- read.csv("mixture.csv")
# 样品数据,包含 3 列,其中一列为分组依据,另外两列为分析使用的代用指标。
mysources <- read.csv("sources.csv")
# 端元信息,至少包含 3 列,分别为端元名称以及两个代用指标的取值。
# 此外,还看过根据需要加上各端元值的标准偏差(不确定度)和 concentration dependence 数据。
simmr_groups <- simmr_load(
mixtures = mydata[,c("d13C", "d15N")], # 读取样品数据表中的代用指标信息
source_names = mysources$Sources, # 读取端元信息表中的端元值名称信息
source_means = mysources[,c("Meand13C", "Meand15N")], # 读取端元信息表中的代用指标取值信息
source_sds = mysources[,c("SDd13C", "SDd15N")], # 读取端元信息表中的代用指标标准偏差(不确定度)信息
group = mydata$group # 读取样品信息表中的分组信息
)
# 2 检查数据
# 绘制数据分布图,查看数据点是否落在所有端元围成的多边形内,以确认端元选取的合理性
plot(simmr_groups) + theme(text = element_text(family = "myfont"))
# 3 启动模型
# 使用 MCMC 模式后需要进行收敛性检验,确保结果合理、有效。
simmr_groups_out <- simmr_mcmc(simmr_groups)
# 也可以直接用 FFVB 模式,则无需进行检验。
# simmr_groups_out_ffvb <- simmr_ffvb(simmr_groups)
# 4 输出结果
# 建议通过 "group = n" 参数设置一下分组,否则要么只显示第1组的计算结果,要么会给出一大堆图像。
# 输出统计分析结果表
summary(simmr_groups_out, type = "statistics", group = 1:max(mydata$group))
# 绘制概率密度分布图
plot(simmr_groups_out, type = "density", group = 1:max(mydata$group))
执行计算程序
进入工作文件夹,终端执行如下命令(Windows、Linux 皆可):
Rscript simmr.R
解读输出结果
执行上述命令后,您会看到一堆命令输出,以及得到一个 Rplot.pdf 文档。
执行脚本后,您可能会得到如下形式的输出:
Summary for 1
mean sd
deviance 6.549 1.321
C3植物 0.145 0.077
海洋POM 0.196 0.124
龙须菜与海带 0.282 0.184
人为污染物 0.376 0.140
sd[d13C] 0.001 0.000
sd[d15N] 0.001 0.000
其中,
Summary for 1表示这是第 1 组数据的分析结果的汇总- 从表格第 2 行开始,每行的第 1 列表示某一来源的名称,第二列是这一来源在物质总量中所占的比例,第三列是这一计算结果的不确定度。
- 比如根据本表,样品所含的总有机碳中,人为污染物来源有机物所占的份额为 37.6±14.0%
接下来我们看一下 Rplots.pdf,这里提供的图表在撰写报告时可能用得上。该文档的第一页大概长这个样子:
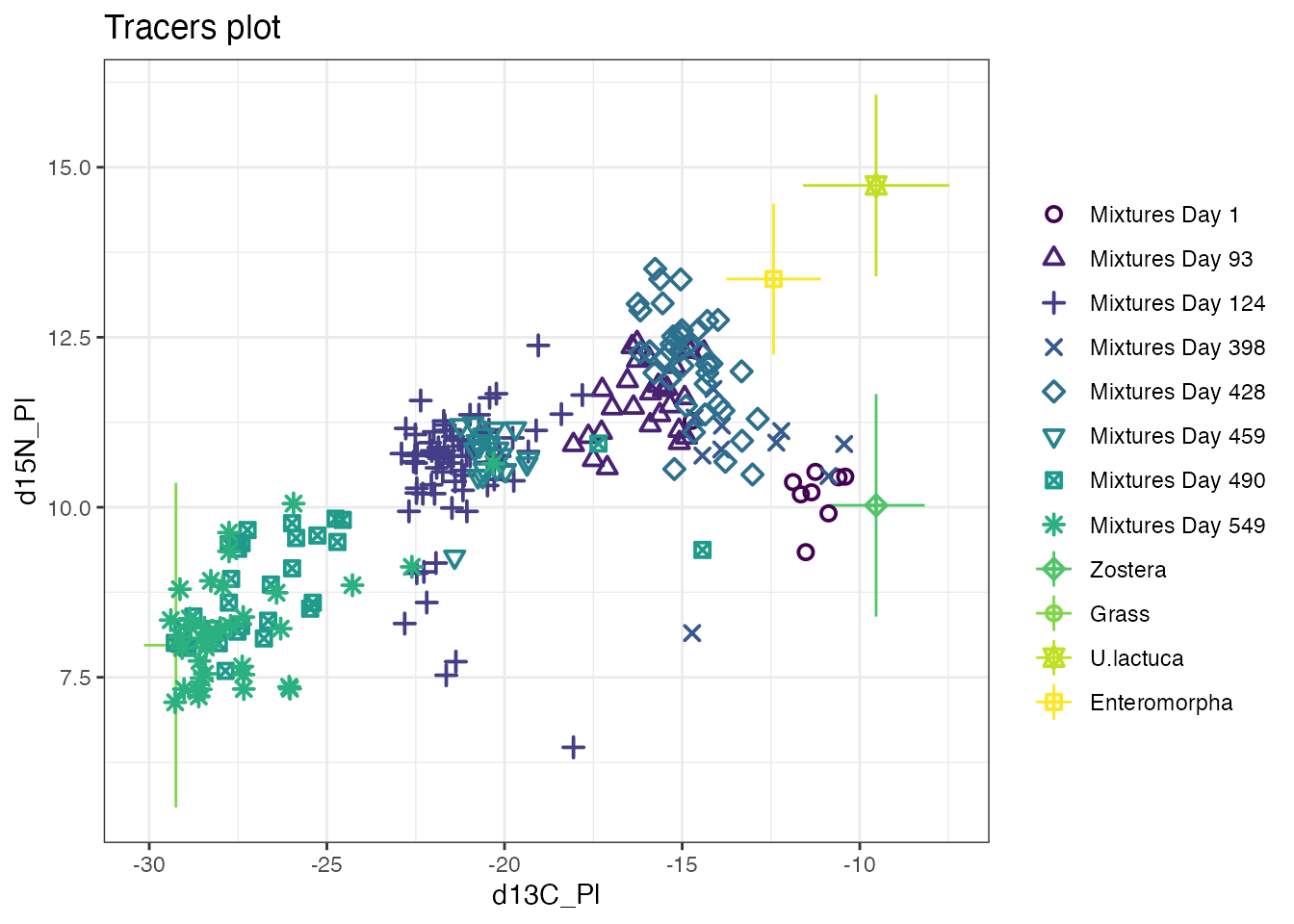
这张图主要是让我们确认端元值选取的合理性。需要注意的是,为得到可靠的结果,所有样品的数据点 必须落在代表各端元值的点围成的凸多边形内。如果有较多的点落在这一区间外,大概率是因为端元的选取不合理,比如忽略了某种重要的物质来源(举个例子,你的样品是在化工厂排污口旁边采集的,但你在设置端元的时候没有包含工业废水)。
接下来的几页大概长这样:

x 轴表示某种来源的物质所占的份额,纵轴表示概率密度。simmr 给出的分析结果都是估算,因此我们只能说这个结果有多大的概率是多少,而不能说这个结果一定是多少。图中的峰值在哪儿,就代表这种来源的物质所占的比例最有可能是多少。
如何在论文中引用 simmr?
simmr 的官网有详细说明:
或者引用这篇发布并介绍该程序的论文: